Bilgisayar Simülasyonları Biyokimya ile Buluşuyor
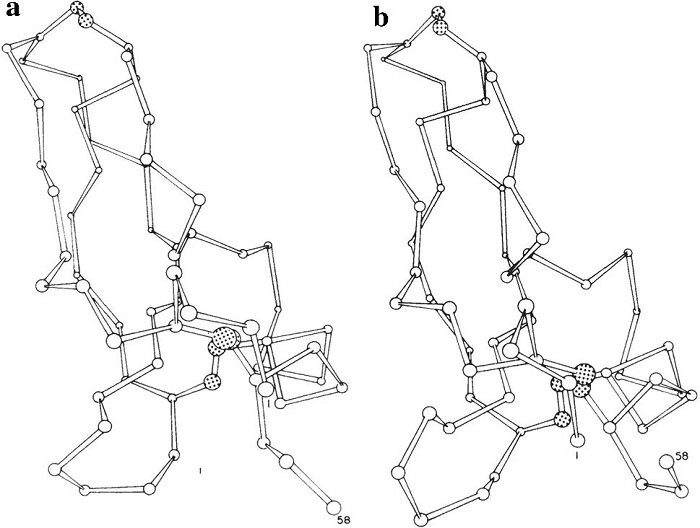
Görsel: BPTI’nin peptit omurgası ve disülfit bağları. a) X-ışını orijinal yapısı; b) 3,2 ps’den sonra gelişen yapı. Her iki gösterimde de alfa karbonları daireler ile, disülfit bağlarındaki sülfürler ise kesikli daireler ile işaretlenmiştir (McCammon ve ark., 1977).
Hayat harekettir. Dolayısı ile canlı organizma fonksiyonlarını anlamak için, onları oluşturan atomların ve moleküllerin hareketlerini ve etkileşimlerini anlamak gerekmektedir. “Moleküler dinamik simülasyonu” adı verilen bu yaklaşım, araştırmacıların, bilgisayarlar aracılığı ile moleküler bir sistemdeki tüm atomların dinamik hareketlerini simüle etmelerini sağlamaktadır.
EPJ Historical Perspectives on Contemporary Physics dergisindeki yeni bir çalışmada, Çin, Pekin Üniversitesi’nden Daniele Macuglia, ABD, Chicago Üniversitesi’nden Benoît Roux ve İtalya, Roma Üniversitesi’nden Giovanni Ciccotti, teorik kimyager Martin Karplus ve ekibinin büyük bir biyolojik molekülün ilk moleküler dinamik simülasyonunu nasıl gerçekleştirdiğini açıklamaktadırlar. Bu molekül, bir protein, 20. ve 21. yüzyıllarda biyoloji ve fizik bilimlerini derinden etkilemiştir.
Şu anda yapay öğrenim araştırmacıları, zamana bağlı hareketlerini ve aralarındaki kuvvetleri yöneten işlevi daha iyi anlamak için biyomoleküler simülasyonlar kullanmaktadırlar.
1970’lerin başında fizikçiler ve fiziksel kimyagerler, soy gazlardan oluşan su ve sıvılar gibi basit maddelerin davranışlarını incelemek adına moleküler dinamik simülasyonunu kullanmaya başlamışlardır. Martin Karplus ve ekibi, bu yöntemi ilk kere büyük bir biyolojik moleküle uygulayarak çalışmada daha da ilerletmişlerdir.
Proteinler, katlanıp bükülerek farklı işlevler kazanabilen minyatür makineler olarak düşünülebilirler. Karmaşıklıkları sebebi ile, zaman içindeki değişimlerini simüle etmek hayli zorludur.
Karplus bu yaklaşımı, bir proteinin ilk moleküler dinamik simülasyonunu gösteren 1977 tarihli bir makale ile duyurmuştur. Günümüze doğru, kimyasal reaksiyonların doğru bir şekilde modellenmesine olan katkısı sayesinde, Arieh Warshel ve Michael Levitt ile birlikte 2013 Nobel Kimya Ödülü’ne layık görülmüştür.
İlgili araştırmanın yazarları, Karplus’un 1977 tarihli makalesinin, hesaplamalı istatistiksel mekaniği biyokimya ile başarılı bir şekilde birleştirmesi ile çalışmalarını mümkün kıldığını belirtmektedirler.
Makaleyi görüntülemek için “Protein dinamiği simülasyonlarının ortaya çıkışı: hesaplamalı istatistiksel mekanik biyokimya ile nasıl tanıştı?” buraya tıklayın.
Kaynak: phys.org.